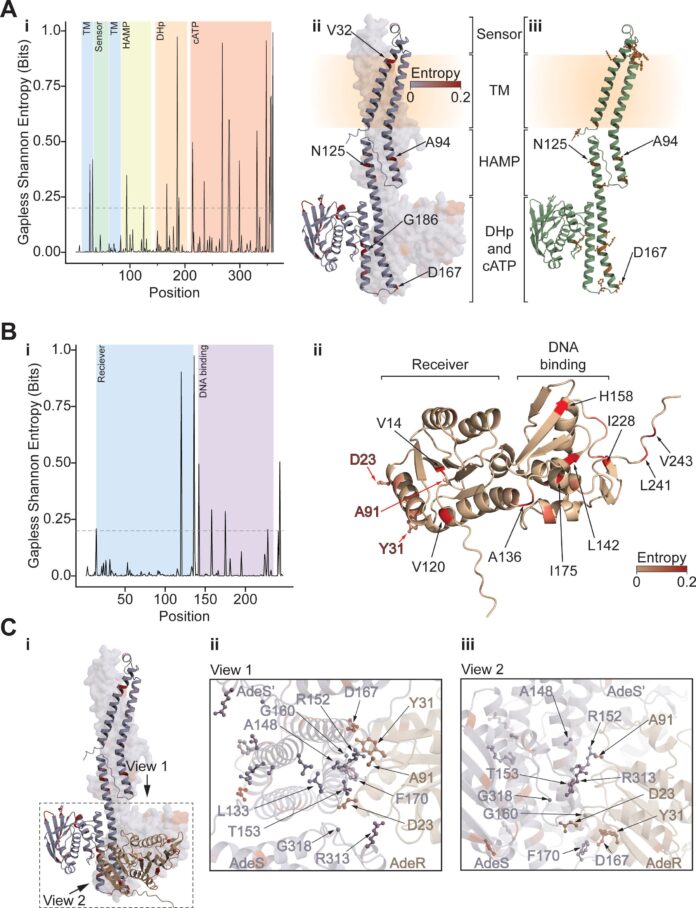
Acinetobacter baumannii (A. baumannii ) stellt eine erhebliche und wachsende Bedrohung für US-Krankenhäuser dar und betrifft mehr als einen von hundert Patienten. Dieses Bakterium ist für sein anpassungsfähiges Genom und die schnelle Entwicklung von Antibiotikaresistenzen berüchtigt, was die Behandlung von Infektionen erschwert und oft schwerwiegende Folgen hat.
Die wachsende Bedrohung durch A. baumannii Infektionen
Die Besorgnis um A. baumannii wird durch Untersuchungen untermauert, die darauf hinweisen, dass etwa ein Drittel der im Krankenhaus erworbenen Infektionen durch dieses Bakterium in den USA eine Resistenz gegen Carbapeneme, eine wichtige Klasse von Antibiotika, aufweisen. Diese resistenten Infektionen sind mit erhöhten Sterblichkeitsraten, längeren Krankenhausaufenthalten und häufigen Verlegungen in andere Gesundheitseinrichtungen verbunden – Ergebnisse, die die Dringlichkeit des Verständnisses und der Bekämpfung dieser bakteriellen Bedrohung verdeutlichen.
Kartierung von Widerstandsmechanismen: Ein experimenteller Evolutionsansatz
Forscher von Sanford Burnham Prebys und Roche Pharmaceuticals haben kürzlich Ergebnisse in Antimicrobial Agents and Chemotherapy veröffentlicht und einen neuen Ansatz zur Kartierung der genetischen Mutationen beschrieben, die die Antibiotikaresistenz bei A auslösen. baumannii. Ihre Methode nutzt eine „experimentelle Evolution“-Technik und konzentriert sich auf zwei ungewöhnliche Antibiotika, Tigecyclin und Colistin – oft als letzte Verteidigungslinie gegen A. baumannii -Infektionen.
„Diese Antibiotika stellen für Ärzte den letzten Ausweg bei A. baumannii -Infektionen dar“, erklärte Andrei Osterman, Ph.D., ein an der Forschung beteiligter Professor. „Während die Resistenz in den USA derzeit relativ gering ist, nimmt sie zu, was uns zu unserer Untersuchung veranlasst, wie diese Bakterien neue Resistenzmechanismen entwickeln.“
Der Morbidostat: Eine bakterielle Evolutionsmaschine
Der innovative Ansatz des Teams nutzt ein spezielles Gerät namens Morbidostat. Dieses System ermöglicht ein kontinuierliches Bakterienwachstum unter zunehmendem Antibiotikadruck über mehrere Generationen hinweg. Ein Computer steuert den Prozess, überwacht die Wachstumsrate der Kultur und führt schrittweise steigende Konzentrationen des Antibiotikums ein, wodurch die Bedingungen im menschlichen Körper effektiv nachgeahmt werden – ein realistischeres Szenario als herkömmliche Labormethoden.
„Betrachten Sie es als eine Evolutionsmaschine“, beschrieb Osterman. „In Kombination mit der Genomsequenzierung können wir eine umfassende Karte praktisch aller möglichen Mutationen erstellen, die Antibiotikaresistenz verleihen.“
Wichtige Ergebnisse: Resistenzpfade für Tigecyclin und Colistin
Die Kartierungsbemühungen der Forscher bestätigten und erweiterten erfolgreich das vorhandene Wissen über die primären Mechanismen der Resistenz gegen Tigecyclin und Colistin. Bei Tigecyclin ist der häufigste Weg zur Resistenz Mutationen, die die Aktivität von Effluxpumpen steigern – Systeme innerhalb der Bakterien, die das Medikament isolieren und entfernen, bevor es die Zelle schädigen kann.
„Dies ist ein gut etablierter Mechanismus“, bemerkte Osterman, „und unsere Ergebnisse erweitern unser Verständnis des Spektrums an Mutationen, die an der Tigecyclin-Resistenz bei A. baumannii beteiligt sind, erheblich.“
Im Hinblick auf die Colistin-Resistenz identifizierte das Team Mutationen, die die Aktivität eines bestimmten Enzyms beeinflussen. Diese Mutationen behindern die Fähigkeit des Enzyms, das Antibiotikum an sein vorgesehenes Ziel innerhalb der Bakterienzellwand zu transportieren.
Prädiktive Genomik: Ein Weg zur gezielten Behandlung
Die Forscher sind optimistisch, dass dieses neu gewonnene Wissen zur Vorhersage von Antibiotikaresistenzen im klinischen Umfeld genutzt werden kann. Durch den Vergleich ihrer umfassenden Karte der Resistenzmutationen mit den Genomen von aus Patienten isolierten Bakterien können sie Vorhersagen über die Arzneimittelanfälligkeit treffen und fundierte Behandlungsentscheidungen treffen.
„Wir haben jetzt Zugriff auf über 10.000 öffentlich zugängliche Genome von A. baumannii -Isolaten, die wir in unsere vergleichende Analyse einbezogen haben“, erklärte Osterman.
Diese Forschung stellt in Kombination mit früheren Studien zur Antibiotikaresistenz bei anderen Krankheitserregern einen bedeutenden Schritt hin zu genombasierten Vorhersagen von Arzneimittelresistenzen dar.
Die Folgen der Versuch-und-Irrtum-Behandlung
Die derzeitige Praxis, Infektionen durch Versuch und Irrtum zu behandeln – Antibiotika zu verschreiben, ohne zu wissen, ob die Bakterien bereits resistent sind – kann die Ausbreitung von Resistenzen unbeabsichtigt beschleunigen. „Wenn Patienten mit Antibiotika behandelt werden, gegen die die Bakterien bereits resistent sind, fördert dies die Resistenz weiter und verschwendet wertvolle Zeit, die den Patienten möglicherweise nicht zur Verfügung steht“, schlussfolgerte Osterman. Die Fähigkeit, Resistenzprofile vorherzusagen, könnte die Herangehensweise von Ärzten an die Behandlung revolutionieren und den Weg für gezieltere und wirksamere Therapien ebnen.