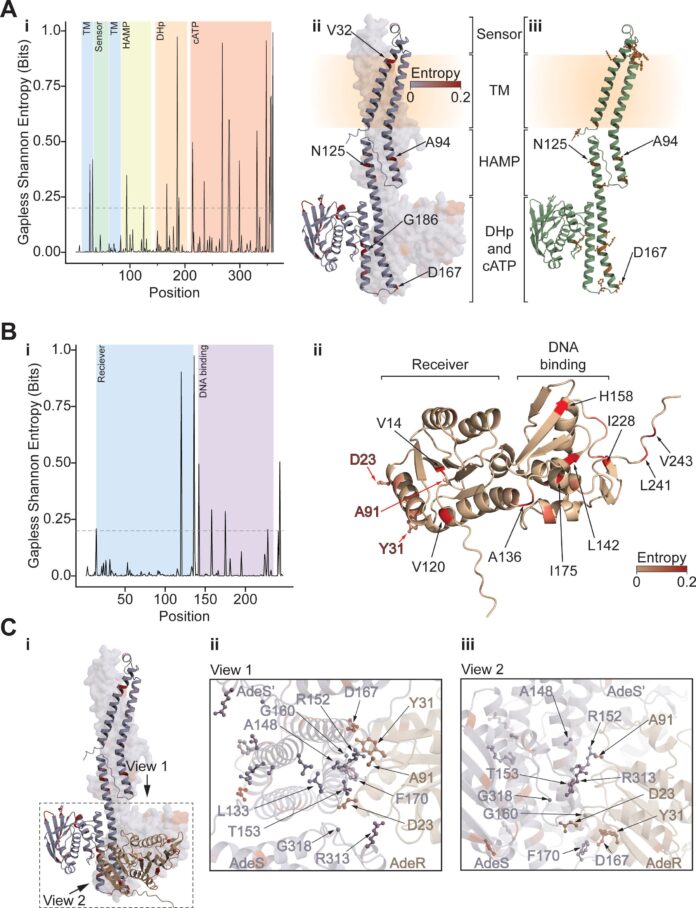
Acinetobacter baumannii (A. baumannii ) plantea una amenaza importante y creciente en los hospitales de EE. UU., que afecta a más de uno de cada cien pacientes. Esta bacteria es conocida por su genoma adaptable y su rápido desarrollo de resistencia a los antibióticos, lo que dificulta el tratamiento de las infecciones y, a menudo, tiene consecuencias graves.
La creciente amenaza de A. baumannii Infecciones
La preocupación que rodea a A. baumannii está respaldado por investigaciones que indican que aproximadamente un tercio de las infecciones hospitalarias por esta bacteria en los EE. UU. demuestran resistencia a los carbapenémicos, una clase crucial de antibióticos. Estas infecciones resistentes se asocian con mayores tasas de mortalidad, estadías hospitalarias prolongadas y traslados frecuentes a otros centros de atención médica, resultados que resaltan la urgencia de comprender y combatir esta amenaza bacteriana.
Mapeo de mecanismos de resistencia: un enfoque de evolución experimental
Investigadores de Sanford Burnham Prebys y Roche Pharmaceuticals publicaron recientemente hallazgos en Antimicrobian Agents and Chemotherapy, detallando un nuevo enfoque para mapear las mutaciones genéticas que impulsan la resistencia a los antibióticos en A. baumannii. Su método utiliza una técnica de “evolución experimental”, centrándose en dos antibióticos poco comunes, tigeciclina y colistina, a menudo considerados defensas de última línea contra A. baumannii infecciones.
“Estos antibióticos representan el último recurso de los médicos para las infecciones por A. baumannii “, explicó Andrei Osterman, Ph.D., profesor involucrado en la investigación. “Si bien la resistencia es actualmente relativamente baja en los EE. UU., está aumentando, lo que impulsó nuestra investigación sobre cómo estas bacterias adquieren nuevos mecanismos de resistencia”.
El morbidostato: una máquina de evolución bacteriana
El enfoque innovador del equipo aprovecha un dispositivo especializado llamado morbidostato. Este sistema permite el crecimiento bacteriano continuo bajo una presión antibiótica cada vez mayor a lo largo de varias generaciones. Una computadora controla el proceso, monitorea la tasa de crecimiento del cultivo e introduce progresivamente concentraciones crecientes del antibiótico, imitando efectivamente las condiciones dentro del cuerpo humano, un escenario más realista que los métodos de laboratorio tradicionales.
“Piense en ello como una máquina de evolución”, describió Osterman. “Combinado con la secuenciación genómica, nos permite construir un mapa completo de prácticamente todas las mutaciones posibles que confieren resistencia a los antibióticos”.
Hallazgos clave: Vías de resistencia a la tigeciclina y la colistina
Los esfuerzos de mapeo de los investigadores confirmaron y ampliaron con éxito el conocimiento existente sobre los mecanismos primarios de resistencia tanto a la tigeciclina como a la colistina. Para la tigeciclina, la ruta más común hacia la resistencia implica mutaciones que mejoran la actividad de las bombas de eflujo, sistemas dentro de las bacterias que aíslan y eliminan el fármaco antes de que pueda dañar la célula.
“Éste es un mecanismo bien establecido”, señaló Osterman, “y nuestros hallazgos amplían significativamente nuestra comprensión de la variedad de mutaciones implicadas en el impulso de la resistencia a la tigeciclina en A. baumannii “.
En cuanto a la resistencia a la colistina, el equipo identificó mutaciones que afectan la actividad de una enzima específica. Estas mutaciones obstaculizan la capacidad de la enzima para administrar el antibiótico a su objetivo previsto dentro de la pared celular bacteriana.
Genómica predictiva: un camino hacia el tratamiento dirigido
Los investigadores son optimistas en cuanto a que este conocimiento recién adquirido pueda utilizarse para predecir la resistencia a los antibióticos en entornos clínicos. Al comparar su mapa completo de mutaciones de resistencia con los genomas de bacterias aisladas de pacientes, pueden hacer predicciones sobre la susceptibilidad a los medicamentos e informar las decisiones de tratamiento.
“Ahora tenemos acceso a más de 10.000 genomas disponibles públicamente de aislamientos de A. baumannii, que hemos incorporado a nuestro análisis comparativo”, explicó Osterman.
Esta investigación, combinada con estudios previos sobre la resistencia a los antibióticos en otros patógenos, representa un paso significativo hacia las predicciones de resistencia a los medicamentos basadas en la genómica.
Las consecuencias del tratamiento de prueba y error
La práctica actual de tratar infecciones mediante prueba y error (prescribir antibióticos sin saber si las bacterias ya son resistentes) puede acelerar inadvertidamente la propagación de la resistencia. “Cuando los pacientes son tratados con antibióticos a los que las bacterias ya son resistentes, se promueve aún más la resistencia y se pierde un tiempo valioso que los pacientes tal vez no tengan”, concluyó Osterman. La capacidad de predecir perfiles de resistencia podría revolucionar la forma en que los médicos abordan el tratamiento, allanando el camino para terapias más específicas y efectivas.